全基因组选择育种简介
育种具有悠久的历史,从一万年前开始,人类就通过表型对动植物进行选择。选择表型优良的亲本,进行杂交或留种,希望保留亲本的优良性状。在分子生物学出现以前,育种家就利用个体之间的亲缘关系,通过数学模型,预测子代的表型。在分子标记开发出来以后,科学家利用基因型信息衡量个体之间的亲缘关系,提升了育种的效率。


图1 神奇的育种帮助我们养活全世界的人口
2001年,Meuwissen等人提出了全基因组选择的概念。全基因组选择育种是一种利用参考群的基因型和表型数据,结合被预测群体的基因型数据对未知表型数据进行预测、选择的重要手段。根据基因组估计育种值(Genomic Estimated Breeding Value, GEBV)进行早期个体的预测和选择,从而缩短世代间隔,加快育种进程,节约大量成本。
全基因组选择育种对低遗传力以及难以测量的复杂性状具有良好的预测效果,真正实现了通过基因组技术来指导育种实践。该技术目前主要侧重于早期预测,因此又称为基因组预测(Genomic Prediction,简称 GP)。
全基因组选择育种需要两方面的数据,一是参考群的基因型和表型数据库,二是预测群的基因型数据。通过数据分析,对预测群的表型进行预测,根据预测打分(估计育种值)对个体进行排序,选择得分高者。
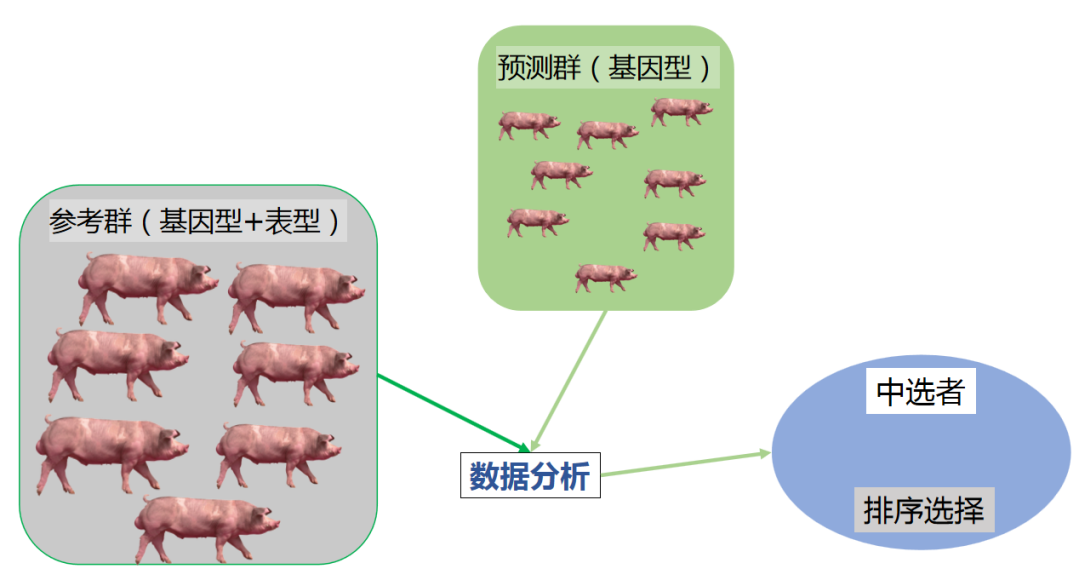
图2 全基因组选择育种流程示意图
今天小欧给大家介绍一篇使用2bRAD简化基因组测序数据,进行凡纳滨对虾饲料利用率的全基因组选择育种的文章。本文在一作解读的基础上整理而成,希望对想做全基因组选择育种的你有所启发。
项目文章
文章:Evaluation of the utility of genomic information to improve genetic evaluation of feed effiffifficiency traits of the Pacifific white shrimp Litopenaeus vannamei
期刊:Aquaculture
发表时间:2020.5.5
作者单位:中国水科院黄海所
文献精读
针对来自34个家系的680尾凡纳滨对虾进行饲料效率测试,每个家系选取20尾对虾,设3个重复,每个重复6~7尾虾,按随机区组设计放入3个循环水养殖系统。测试4周后,去除测试过程中死亡的对虾个体、测试数据不准确的对虾个体、剩余个体数量小于6尾的家系,最后获取519尾对虾的饲料效率性状相关数据,包括剩余摄食量(RFI)、饲料效率比(FER)和平均日增重(ADG)。

图3 虾的养殖与数据收集
从519尾测试个体中挑选了479尾保存较好的个体进行肌肉组织取样,利用2b-RAD简化基因组测序的方法对479个样品进行分型。选择凡纳滨对虾为参考基因组进行电子酶切预测,最终确定使用 BsaXI酶进行实验。对基因组进行电子酶切,预测可获得 773,610 个标签,其中unique标签 675,151 个。各样品去除测序深度小于3的Unique标签后,平均Unique标签数目为 307,641 ,所有样品共获得SNP标记 114,118 个。对所有标记和样本进行质量控制,剔除所有样品中低于80%个体可以分型的位点,剔除MAF低于0.05的位点,剔除只含有1种基因型的位点。质控后保留474个样本和35,869个SNP标记,利用Beagle软件对个体缺失位点进行填充。
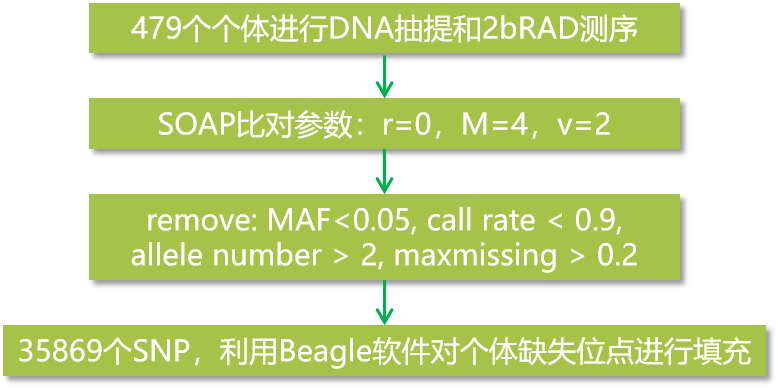
图4 2bRAD测序数据分析
为了提高评估效率和准确性,引进基于SNP信息辅助遗传评估——基因组BLUP(GBLUP)和一步法基因组BLUP(ssGBLUP)。基于BLUPF90软件平台,利用35,869个SNP标记构建474尾分型个体间的分子遗传关系矩阵(G matrix),根据物理系谱获得亲缘关系矩阵(A matrix),将G矩阵和A矩阵进行复合以获得H矩阵。3个矩阵中的成对个体间亲缘系数热图见图5,许多在系谱中不存在亲缘关系的个体依据基因组关系矩阵会存在或近或远的亲缘关系。系谱中家系内两个个体间的亲缘关系系数完全一致,而在G矩阵和H矩阵中明显不同。分型个体在G矩阵和A矩阵中下三角元素间的皮尔森相关系数高达0.92。3个矩阵中下三角元素的分布见图6。总体上,大部分下三角元素都在0附近。A矩阵中的下三角元素分布从0到0.563,仅有29个数值。相比之下,G矩阵(-0.122~0.636)和H矩阵(-0.074~0.652)的下三角元素的分布变异更大。

图5 三个矩阵的成对个体间亲缘系数热图
横纵坐标均为一个样品,数值关于对角线对称。样品按照家系排序,因此对角线附近为同一家系内的个体,亲缘关系更近。

图6 三个矩阵的下三角元素分布。横坐标是亲缘系数,纵坐标是频率
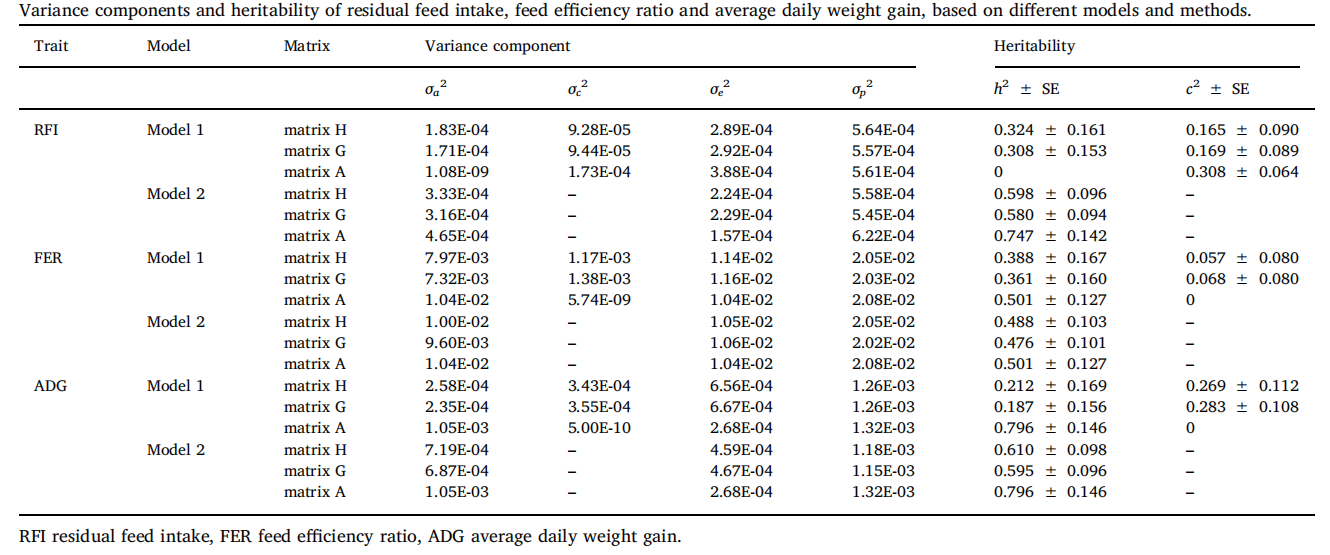
选择养殖系统、日龄、共同环境效应等固定和随机效应因子,建立精确育种分析模型,分别基于A矩阵、G矩阵和H矩阵进行RFI、FER和ADG的方差组分估计,并计算各性状的遗传力,具体采用ASReml 软件基于动物模型和AIREML方法。基于不同矩阵及不同模型估计得到的RFI、FER和ADG方差组分和遗传力见表1。基于系谱的模型无法估计出任何一个性状的共同环境效应,三个性状的遗传力估计值偏高(0.501±0.127~0.796±0.146)。当采用GBLUP和ssGBLUP模型时,三个性状表现出相对较低的遗传力估计值(0.187±0.156~0.388±0.167)以及不同程度的共同环境效应(0.057±0.080~0.283±0.108)。三个性状基于GBLUP模型估计的遗传力相比基于ssGBLUP模型估计的遗传力下降约4.9~11.8%。
表1 基于不同矩阵及不同模型估计得到的RFI、FER和ADG方差组分和遗传力
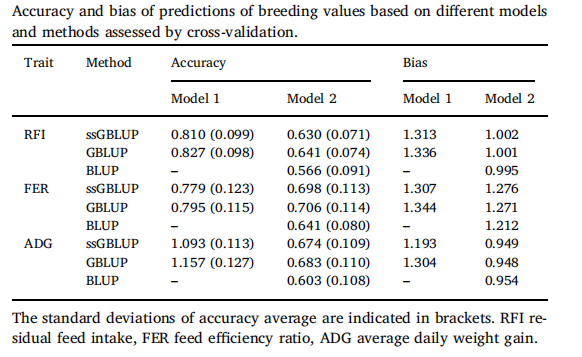
利用5倍的交叉验证评估不同模型估计育种值的准确性以及偏差,准确性具体用候选群体内每个个体的育种值与其观测表型间的皮尔森相关系数除以遗传力的开方表示,模型的偏差是以观测表型对预测表型的回归系数作为度量。不同模型和方法的育种值预测准确性和偏差见表2。当模型中不包括共同环境效应时,基于GBLUP和ssGBLUP估计的各性状基因组育种值准确性为0.716~0.782,明显高于基于BLUP估计的各性状育种值准确性(0.641~0.674)。此外,基于GBLUP估计的各性状基因组育种值准确性要比基于ssGBLUP估计的各性状基因组育种值准确性有所提高。值得注意的是,当模型中包含共同环境效应时估计的基因组育种值准确性要高于模型中不包含共同环境效应时的育种值准确性。
表2 利用交叉验证评估EBV的准确性和偏差
参考资料
介绍部分的图片取自华中农业大学刘小磊老师的课件。










评论