产品详情
文献和实验
相关推荐
DAP-seq(DNA affinity purification sequencing)
产品介绍
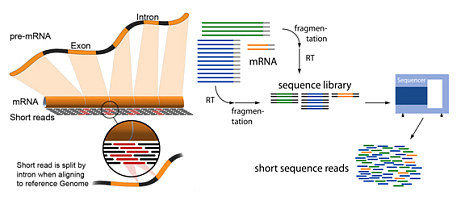
DAP-Seq是将蛋白质体外表达技术与高通量测序技术相结合,使用体外表达的TF蛋白对裸露的全基因组DNA片段进行孵育,以确定结合序列。
通过提取基因组DNA,进行基因组DNA文库构建。随后利用HaloTag载体,将目的蛋白与HaloTag融合表达,将纯化的融合蛋白与基因组文库进行孵育,利用目的蛋白结合DNA片段,并且利用HaloTag特异性磁珠,将目的蛋白和DNA复合物进行提取与纯化,随后对捕获的DNA片段进行扩增,测序分析,探究目的蛋白结合的DNA片段。
通过提取基因组DNA,进行基因组DNA文库构建。随后利用HaloTag载体,将目的蛋白与HaloTag融合表达,将纯化的融合蛋白与基因组文库进行孵育,利用目的蛋白结合DNA片段,并且利用HaloTag特异性磁珠,将目的蛋白和DNA复合物进行提取与纯化,随后对捕获的DNA片段进行扩增,测序分析,探究目的蛋白结合的DNA片段。
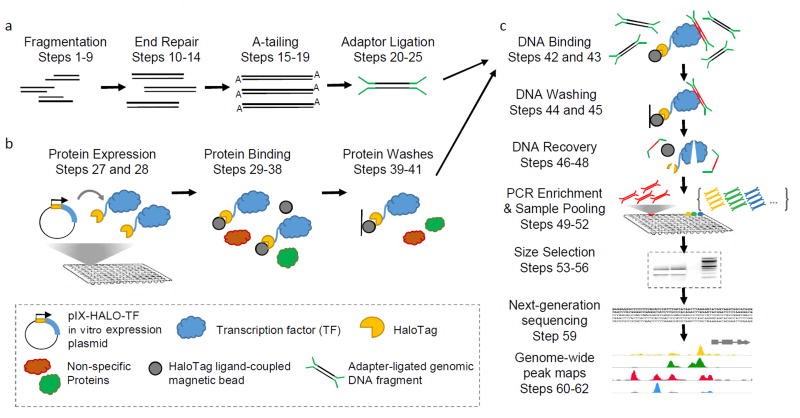
DAP-seq实验流程
技术路线

样本要求:
细胞:>5×104个,
动物组织:≥0.2 g
植物组织:≥0.5 g
真核微生物:500 mg
建库测序:
测序策略:Illumina Hiseq, PE150
测序深度:6-12 G
项目周期:
90个工作
细胞:>5×104个,
动物组织:≥0.2 g
植物组织:≥0.5 g
真核微生物:500 mg
建库测序:
测序策略:Illumina Hiseq, PE150
测序深度:6-12 G
项目周期:
90个工作
《玉米生长素响应因子家族的DNA结合景观》
The DNA binding landscape of the maize AUXIN RESPONSE FACTOR family. Nature communications,2018(IF=11.878)
研究背景
生长素响应因子(AUXIN RESPONSE FACTORS ,ARFs)是植物特异的转录因子,对植物感性生长素调控下游基因表达具有十分重要的作用。与许多大型TF家族一样,一个关键问题是个体成员是否通过结合不同的靶基因来确定发育特异性。
研究结果
通过对A和B两个不同分化枝的共14个ARFs进行DAP-seq分析,发现统一进化枝下的ARFs结合位点具有较强一致性,但是不同进化枝的ARFs结合位点一致性较差。通过结合QTLs分析发现,生长素响应的特异性可能是单个ARF结合位点之外的其他因素决定的。

参考文献
[1] Omalley R C, Huang S C, Song L, et al. Cistrome and epicistrome features shape the regulatory DNA landscape[J]. Cell, 2016, 165(5): 1280-1292.
[2] Bartlett A, Omalley R C, Huang S C, et al. Mapping genome-wide transcription-factor binding sites using DAP-seq[J]. Nature Protocols, 2017, 12(8): 1659-1672.
[3] Stigliani A, Martinarevalillo R, Lucas J, et al. Capturing Auxin Response Factors Syntax Using DNA Binding Models[J]. Molecular Plant, 2019, 12(6): 822-832.

武汉爱基百客生物科技有限公司
实名认证
钻石会员
入驻年限:9年