产品详情
文献和实验
相关推荐
CUT & RUN(cleavage under targets and release using nuclease),即核酸酶靶向切割和释放技术,是一项用于在细胞天然染色质环境下检测蛋白-DNA复合体的新方法,与传统的ChIP-Seq研究方法相比,该技术无需甲醛进行交联剂免疫沉淀等操作,因此具有快速、所需低细胞量,背景信号低、重复性好等优点,可用于单细胞水平研究。CUT & RUN对于表观遗传、基因调控等领域的研究具有革命性意义。
技术原理
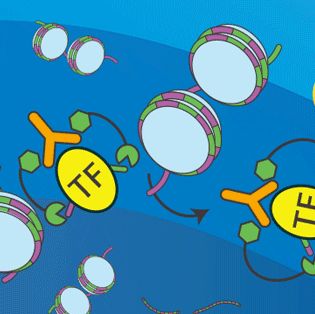
无需用甲醛进行交联剂免疫沉淀,通过针对靶蛋白(如转录因子、染色质重塑蛋白)特异性抗体和特异结合免疫球蛋白G(Protein A)的介导,使与Protein A融合的pAG-微球菌核酸酶(pAG-MNase)切割并释放靶蛋白结合的序列,获得目的DNA片段,并进一步建库测序。
技术应用
1. 挖掘蛋白质—DNA相互作用2. 检测基因组上的组蛋白修饰,鉴定超级增强子3. 检测基因组的开放性、转录因子结合图谱,鉴定超级增强子4. 绘制核小体、RNAPII等蛋白的染色质图谱
技术特点
1. 样本量仅需50万个细胞2. 数据信噪比高,背景低,数据重复性好3. 操作简便,实验操作时间短,仅1天分析内容
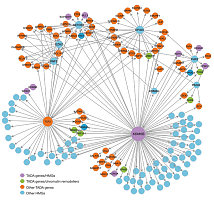
1. 基因组比对分析2. 基因组富集分析2.1 富集区域基本信息2.2 富集区域分布特征2.3 Peak相关基因GO分析2.4 Peak相关基因KEGG分析 2.5 差异Peak相关基因GO,KEGG分析 3. 富集区域motif分析4. 超级增强子分析(H3K27ac抗体、BRD4抗体)
送样要求
样本类型:活细胞,5 x105 个细胞/样本样本物种:仅限人、大小鼠,其他物种需评估。
表观生物实测数据
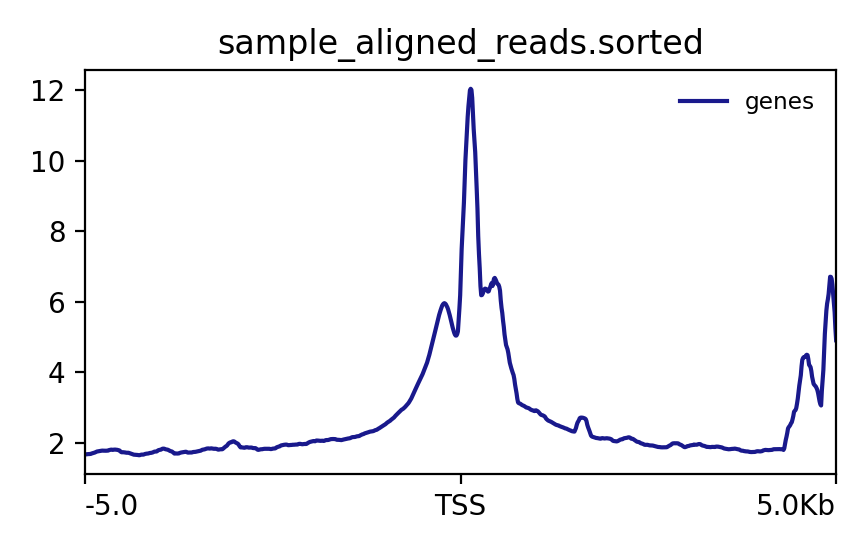
图1. TSS附近的reads分布图
*注释:reads分布在TSS附近
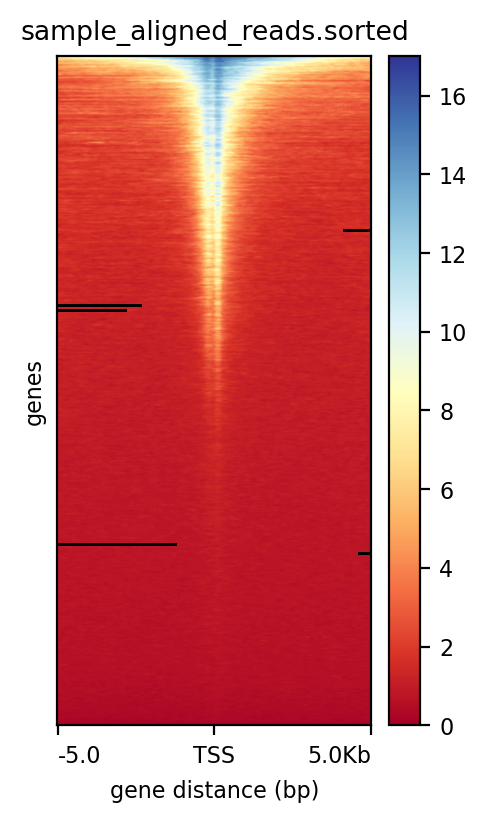
图2 TSS附近的heatmap图
*注释:由热图可见,TSS附近reads富集程度
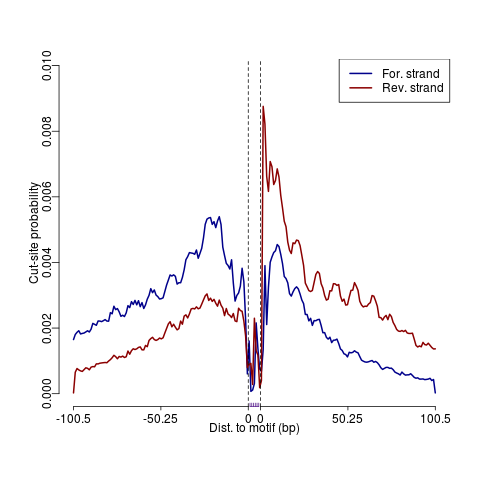
图3 footprinting图
*注释:motif的footprinting可查看motif在各个位置上的切割频率,与峰调用相比,该方法可实现结合位置的更高分辨率映射。切割位点源自通过募集至抗体结合位点的pA-MN融合物染色质切割后产生的单个DNA片段的两端。由于染色质相关的蛋白结合,切割频率较低的区域倾向于受到保护,而没有结合的侧翼区域则显示较高的切割频率,特征性双峰,中心位置反映出motif位置范围
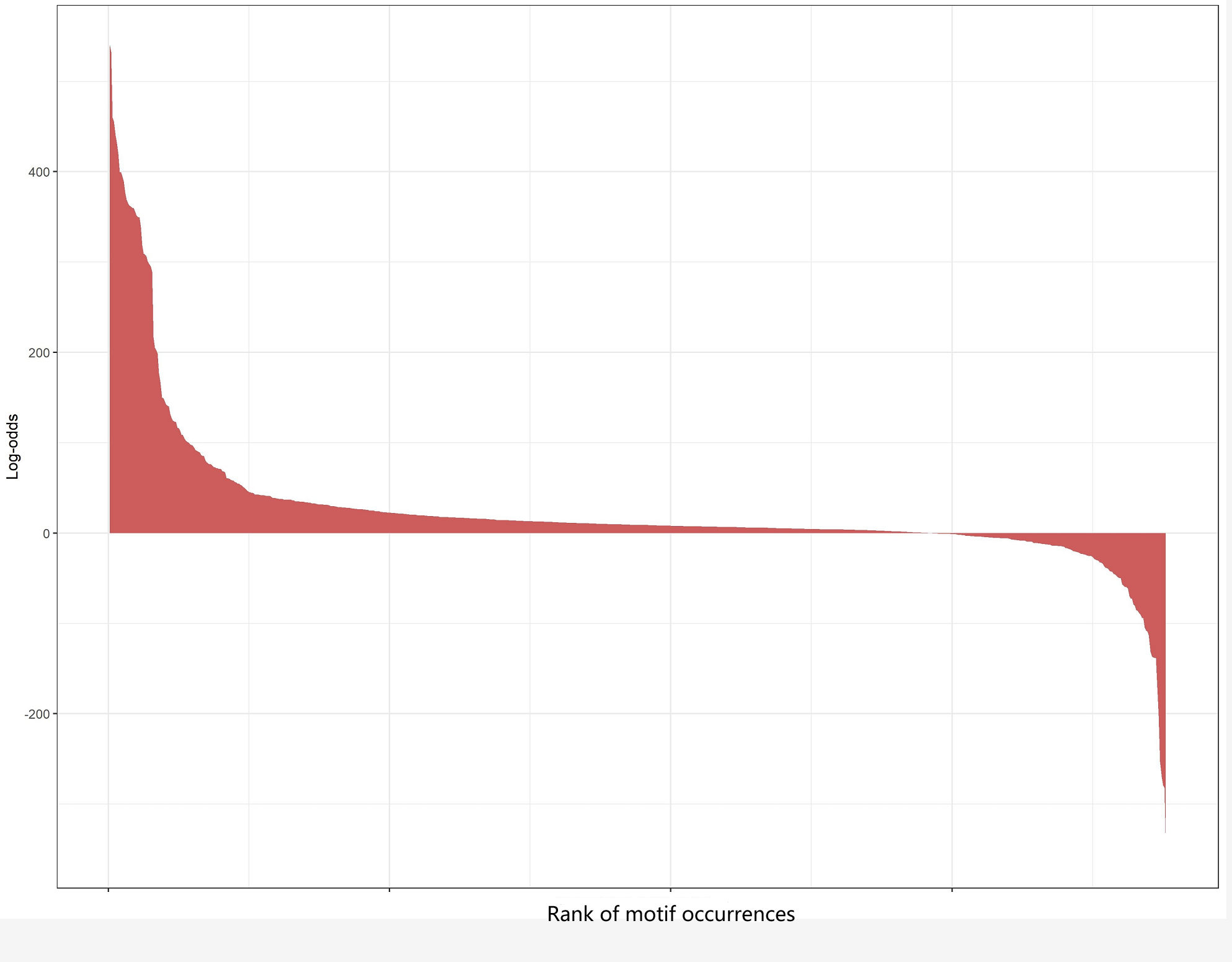
图4:log-odds图
*注释:由log-odds值对数量化了footprinting图中的切割位点,通过log-odds值得分对切割点进行排名,log-odds>5的得分位点可视为直接结合位点。
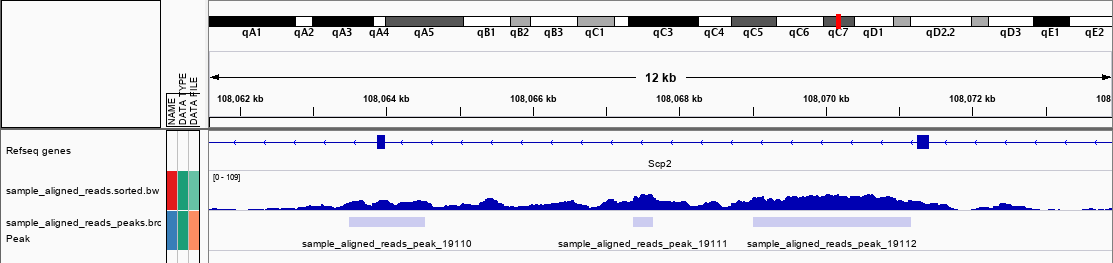
图5:IGV视图
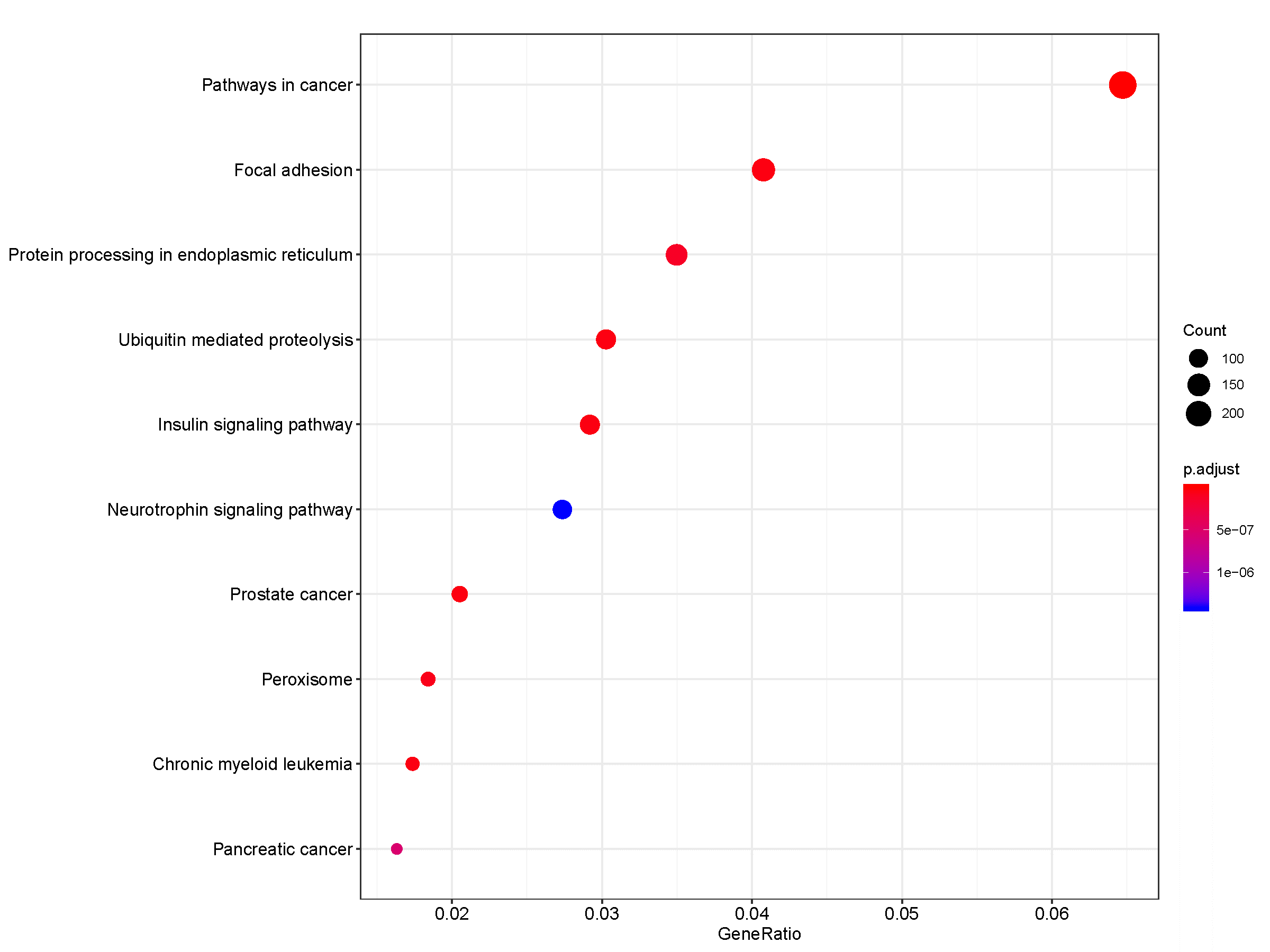
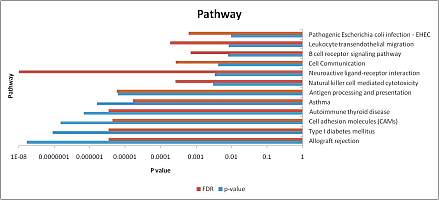
图6:KEGG富集
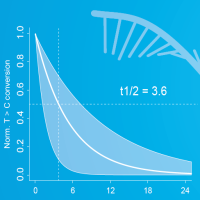
参考案例Cell:CUT & RUN技术的低细胞和高灵敏度的表观基因组学[1]本研究方法检测多个转录因子(干细胞相关转录因子OCT4、SOX2和NANOG)、组蛋白修饰、鉴定超级增强子、染色质的开放性效果。CUT & RUN技术将蛋白质-DNA相互作用应用于低细胞量研究;甚至在单细胞研究中,仍然有较高的灵敏度。1. 低细胞用量:CUT & RUN技术分析CTCF 或H3K4me3测序结果:在这两种蛋白已知结合位点处的read可视化,显示10个细胞起始量的测序结果可以展现出类似50万个细胞量的结合位点富集热图。CTCF的结合区更为狭窄集中,而H3K4me3则占据相对宽的区段。而对照组(无抗体组)的热图则没有这种特征。
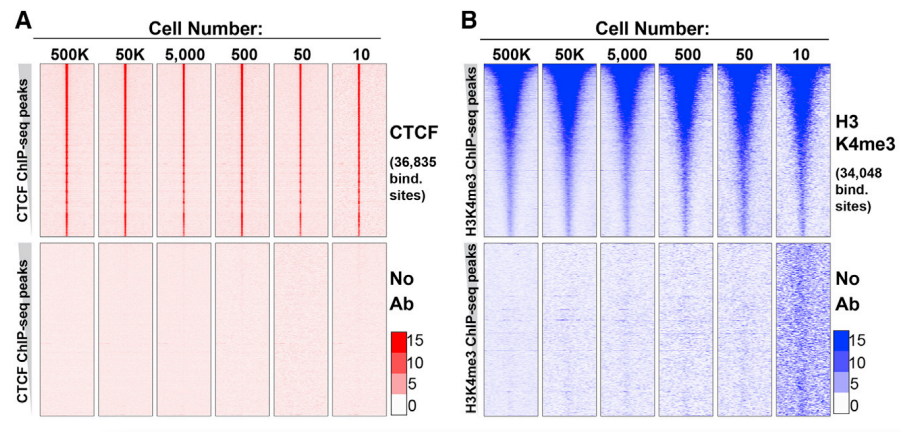
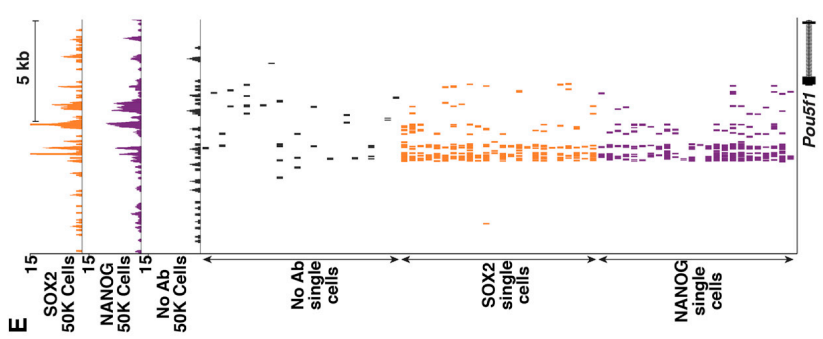
参考文献[1] Sarah J, Ana B, Kurtis N, etal. Profiling of Pluripotency Factors in Single Cells and Early Embryos. Cell,2019, May 16; 177: 1-11.

广州表观生物科技有限公司
实名认证
钻石会员
入驻年限:9年