产品详情
文献和实验
研选推荐
提供商 :康测科技
服务名称 :MeRIP-seq/测序 M6A-seq | 核酸-蛋白互作研究
具体产品价格以及送样要求详细请咨询张经理:13871149015
技术问题可来电咨询:027-65527552
更多表观遗传研究产品——ChIP-seq(详情点击)
MeRIP-seq (RNA甲基化测序)
m6A修饰
m6A的发生、发展
RNA上目前发现存在着100多种不同类型的转录后修饰,并且所有已知的RNA种类都可以被修饰,其中rRNA和tRNA的修饰最多。mRNA上的主要修饰类型包括m6A、m1A和m5C。其中m6A是最丰富的mRNA修饰,也是被研究的最清楚的RNA修饰。m6A修饰存在于mRNA、lincRNA、pri-miRNA和rRNA中。m6A修饰是一个可逆的过程,对m6A进行研究绕不开催化/擦除m6A修饰的修饰酶(Writers)/去修饰酶(Erasers),以及各种阅读器蛋白(Readers),这些蛋白是m6A发生、发展,以及形式其调控功能的最重要的调控因素和效应分子:
Writers:
- METTL3–METTL14复合物是大部分mRNA、lncRNA和miRNA上的m6A修饰的催化酶;
- METTL16则催化负责SAM合成的特定mRNA,和U6 snRNA的甲基化;
- METTL5-TRMT112复合物负责催化18S rRNA的m6A甲基化;
- ZCCHC4负责催化28S rRNA的m6A甲基化;
Erasers:
- ALKBH5是主要的去甲基化酶;
- FTO可能主要负责m6Am的去甲基化;
Readers:
- YTH家族的五个成员,这些蛋白可以通过YTH结构域直接与mRNA上的m6A结合;
- IGF2BP,HNRNPA2B1和PRRC2A等,这些阅读器并不直接识别m6A修饰,而可能是与m6A的诱导的未折叠RNA结合;
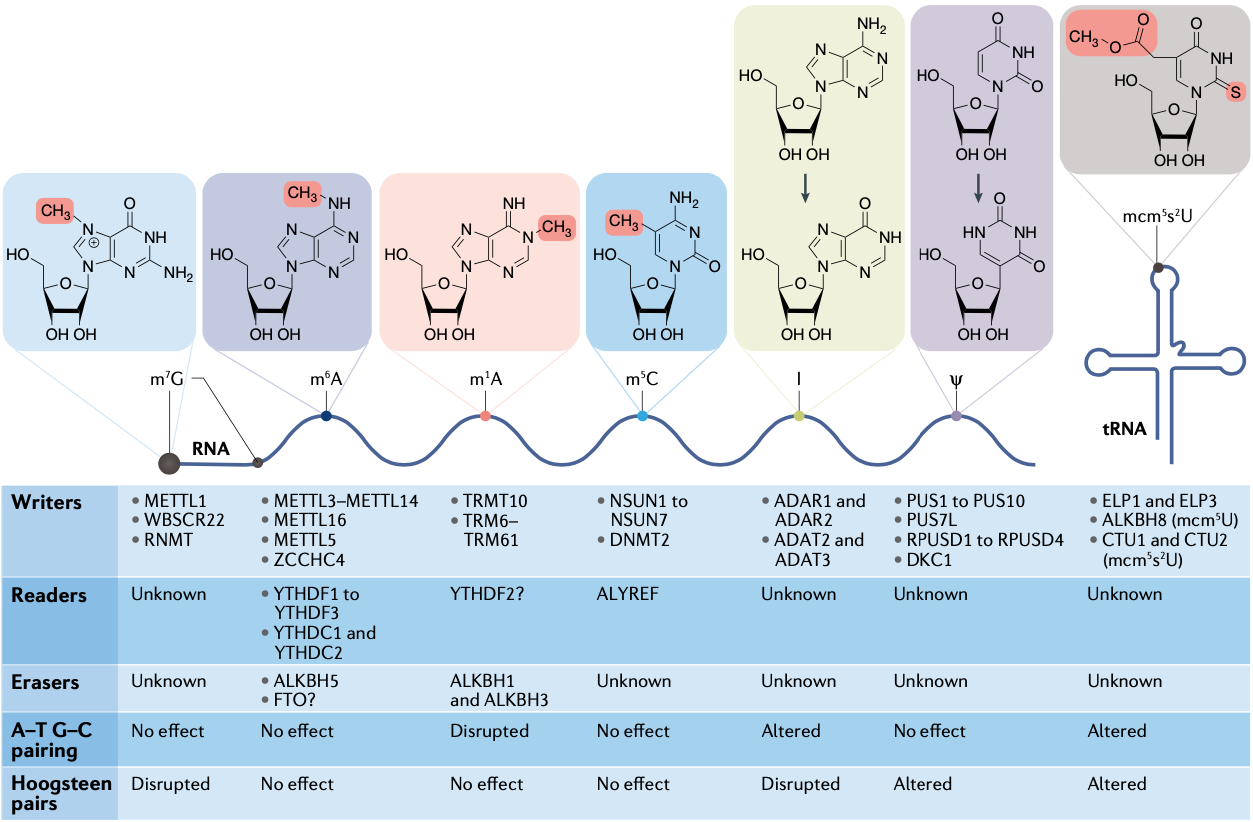
m6A的调控及其功能
m6A修饰目前发现在发育、增殖、分化、癌症发生等很多生物学过程中起着重要的调控作用。m6A修饰在mRNA的5'UTR和CDS的终止密码子附近富集,影响mRNA的稳定性,剪接和翻译,影响miRNA的加工成熟,并影响lincRNA的功能。这些不同的调控功能,重要是通过不同的效应蛋白结合行使的,如YTHDF2介导mRNA降解,而YTHDF1促进mRNA翻译。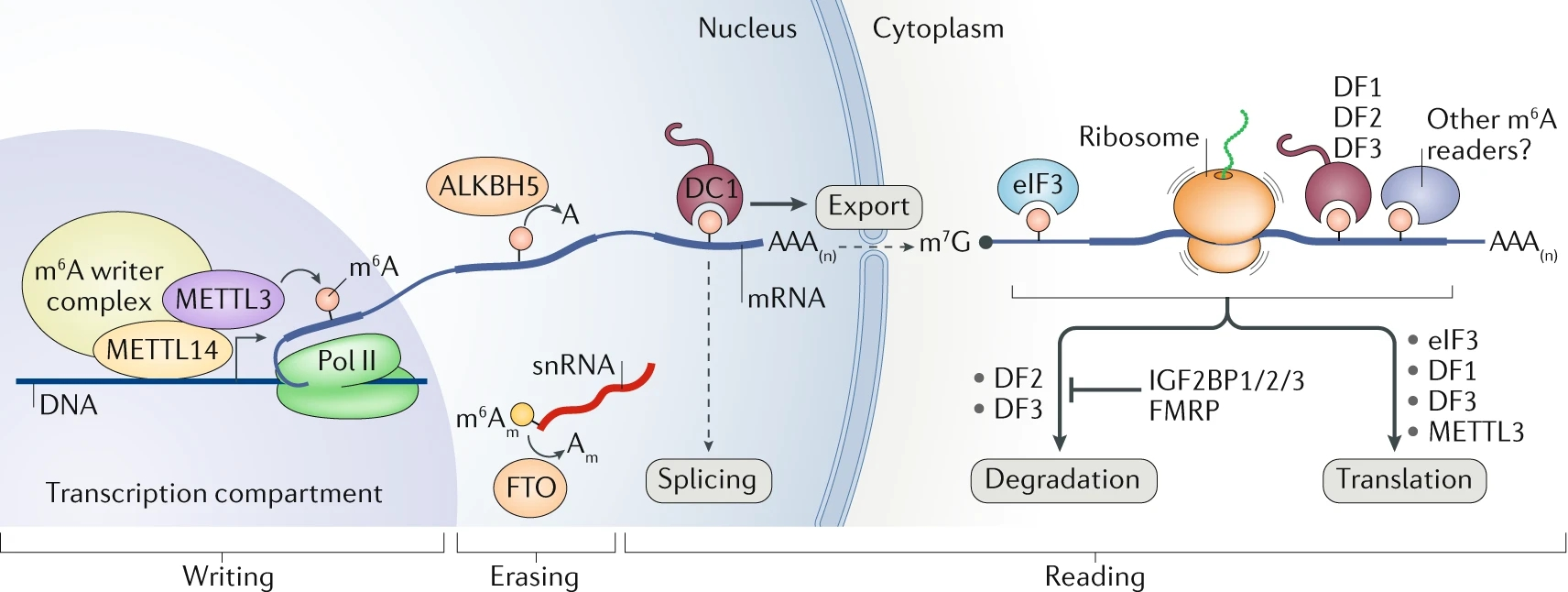
meRIP-seq
- m6A修饰(m6A-seq)
- m1A修饰(m1A-seq)
- m5C修饰(m5C-seq)
meRIP-seq的实验原理如下图所示:首先通过polyA捕获mRNA,或者通过rRNA剔除去掉rRNA,然后将获得的RNA打成100nt左右的小片段;之后使用特定修饰特异的抗体(以m6A抗体为例)进行免疫共沉淀,含有m6A修饰的RNA片段被富集并回收;进而对回收的RNA进行文库构建、高通量测序和生物信息学分析,通过peak分析鉴定出m6A修饰的位点。
由于rRNA中含有m6A修饰,因此实验过程中rRNA的剔除是必需的。目前存在两类技术去除rRNA的干扰,一种是polyA捕获正筛mRNA、另一种是rRNA剔除负筛rRNA,这两种方法成本上存在着巨大的差异,同时其检测对象和结果也显著不同:
- mRNA捕获:便宜,但仅能对mRNA和部分带有polyA尾的lncRNA上的m6A修饰进行研究,是目前最常用的方法;
- rRNA剔除:贵,且受物种限制,但该方法可以全面检测mRNA、lncRNA、circRNA和pre-mRNA上的m6A修饰,是非常全面的方法;
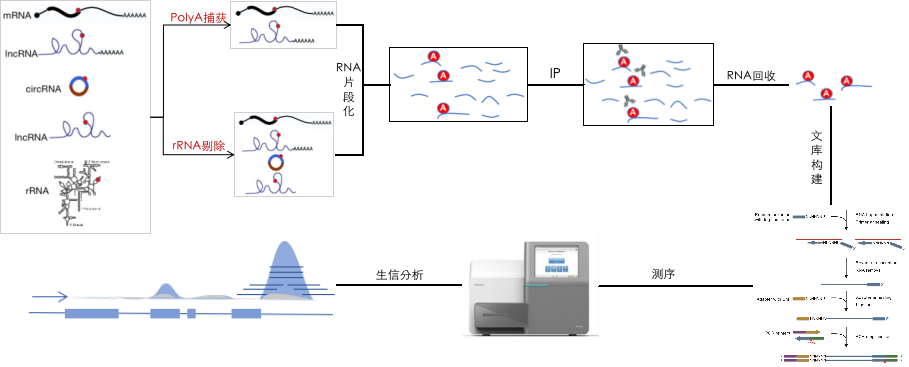
nano meRIP
上述meRIP-seq可以解决大部分m6A修饰研究需求,但是上述方法对RNA的需求量比较大:
- 基于polyA捕获的meRIP/m6A-seq需要20 ug RNA;
- 基于rRNA剔除的meRIP/m6A-seq需要10 ug RNA;
但是对于一些特殊样本,如少量原代细胞、珍贵的临床样本,则可能无法获得足量的RNA,因此无法通过上述meRIP-seq来对m6A修饰进行研究。为了解决这个问题,nano meRIP-seq问世了!
该技术应用了我公司应用在lncRNA-seq和互作转录组中的DSN rRNA剔除方法,实验流程如下:
- 首先将Total RNA进行片段化,至100 nt长度;
- 使用m6A抗体进行IP,并回收RNA;
- 将IP下来的RNA全部用于文库构建:由于rRNA上带有m6A修饰,因此该RNA中含有大量的rRNA,大幅提高了RNA量,使文库构建得以成功;
- 将构建好的文库,使用DSN消化高丰度的rRNA来源的DNA,从而富集mRNA、lncRNA和circRNA;
- 高通量测序和生物信息学分析
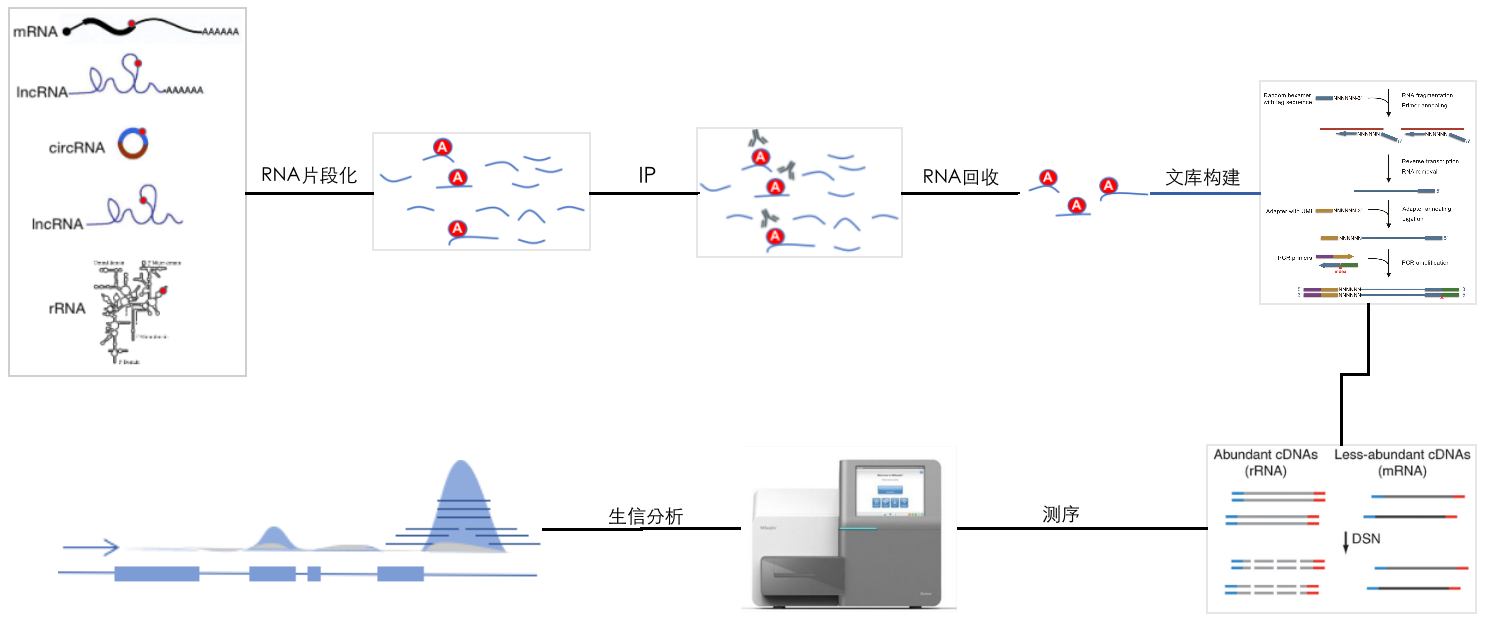
本技术可以针对低至500 ng的Total RNA进行meRIP-seq研究,同时还可以检测lncRNA、circRNA甚至是病毒RNA的的m6A修饰情况。
服务流程
您只需要提供足量的组织/细胞或RNA,我们将根据物种、RNA起始量为您推荐合适的meRIP实验方案~
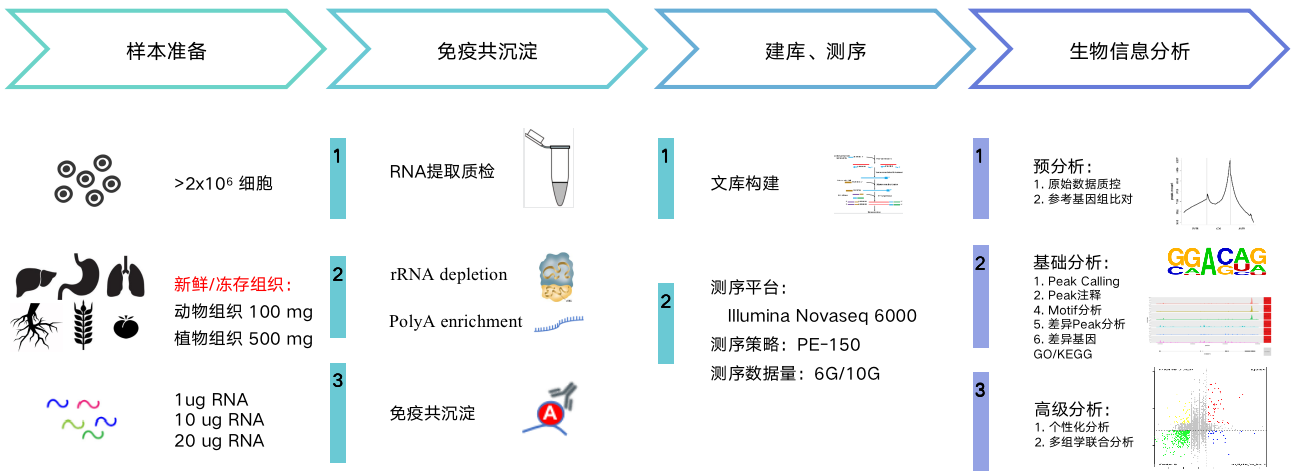
产品优势
数百个项目经验!
UMI RNA-seq建库加成!
国内唯1的高通量、自动化meRIP工作站!

实测数据
我们已对数十个物种,提供了数百个项目的meRIP-seq,积累了丰富的项目经验。以下展示部分核心结果:
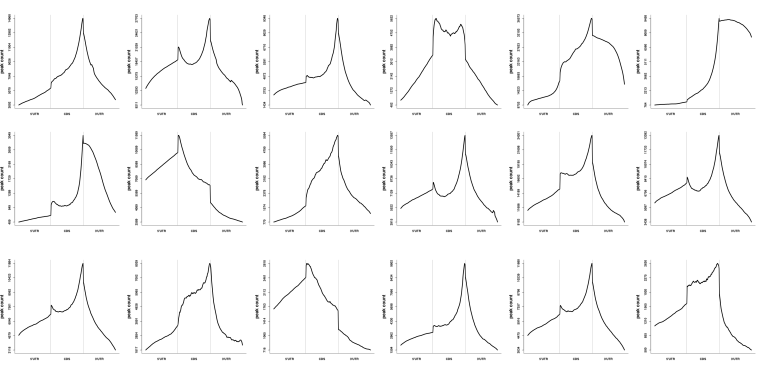
Peak在RNA上的分布
前期的研究表明,哺乳动物的m6A修饰在5‘UTR和stop codon处有富集,随着研究物种、组织类型越来越多,我们发现m6A修饰的特征远远超出我们之前的认识,从下图可以看到:
- stop codon处的富集在大多数物种、大多数组织类型中都是保守的;
- 5’UTR的富集并不明显;
- Start codon处在某些物种中存在显著富集;
- 部分RNA同时在start codon和stop codon处有富集;
- 部分RNA在CDS区有富集;
- 部分RNA在3‘ UTR存在显著富集;
- 同一物种的不同类型,其m6A修饰分布的情况差异很大;
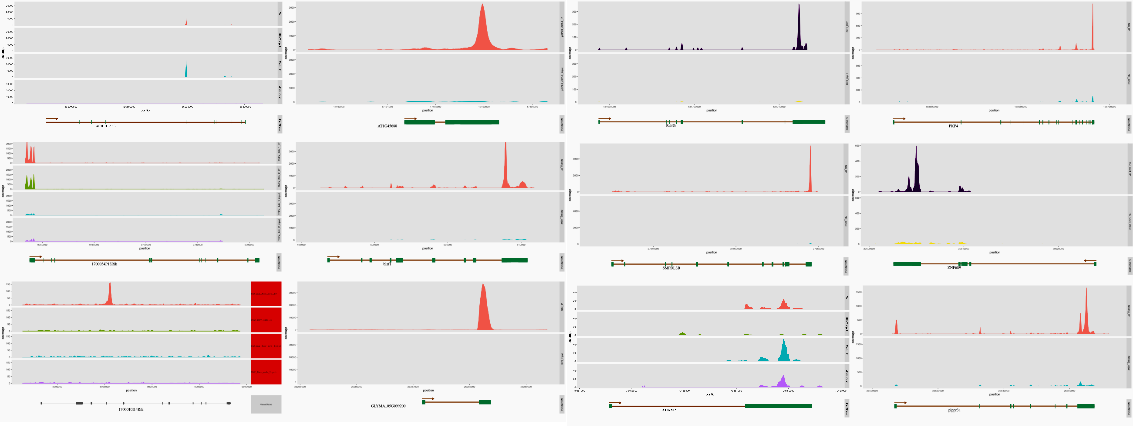
Peak Reads在基因上的分布
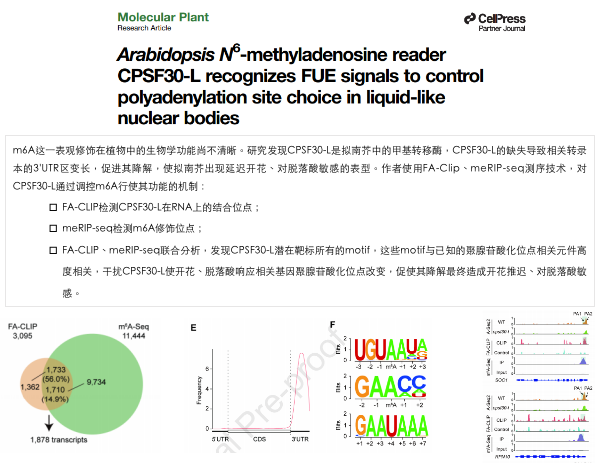
部分客户发表文章(UID-MeRIP)
- Peizhe Song, Junbo Yang, Chunling Wang, Qiang Lu, Linqing Shi, Subiding Tayier, Guifang Jia, Arabidopsis N6-methyladenosine reader CPSF30-L recognizes FUE signal to control polyadenylation site choice in liquid-like nuclear body, Molecular Plant, 2021. (IF:12.812)
- Hou Y , Sun J , Wu B , et al. CPSF30-L-mediated recognition of mRNA m6A modification controls alternative polyadenylation of nitrate signaling-related gene transcripts in Arabidopsis[J]. Molecular Plant, 2021, 14(4).(IF:12.812)

武汉康测科技有限公司
实名认证
钻石会员
入驻年限:7年