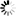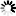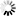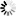
产品详情
文献和实验
相关推荐
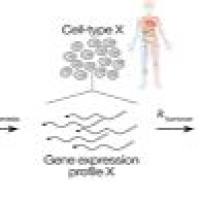
转录组分析的核心目标是准确定量样品中RNA转录本的丰度。
RNA-seq的流程为:RNA片段化→cDNA合成→加接头→PCR扩增→测序。但由于在进行RNA测序时,逆转录、加接头、PCR文库扩增、测序序列依赖性偏差和扩增噪声等原因,使得常规RNA-Seq的结果准确度下降。

2012年PNAS报道了一种可以降低RNA-Seq偏倚和噪音的方法,也是我们常说的UMI-RNA-Seq。

RNA测序在文库构建时,都需要通过PCR扩增,产生重复的分子片段,但是所有的文库片段并非以同等速率等量的扩增,扩增速率受到片段长度、GC含量、片段浓度等多方面的影响,容易扩增的片段被极大的富集,一些含量较低的片段或碱基偏好严重的片段甚至完全丢失,最终影响测序结果的准确性。
UMI-RNA-Seq是在每个cDNA分子扩增之前连接特异序列标签(UMI,Unique Molecular Identifier),这些标签(UMI)会随cDNA序列一起扩增测序。测序完成后,通过识别UMI标签,将具有相同数字标签的重复片段合并,消除PCR扩增中的偏好性(去除PCR扩增及测序仪引起的重复),就可以定量原始样本中cDNA分子的数量。

UMI消除PCR扩增偏好,但UMI≠绝对定量测序!
相比常规转录组测序,通过添加UMI特异性标签,转录本测序定量的准确性确实能够实现大幅提高,但通过UMI-RNA-Seq就能真正的实现绝对定量测序吗?
根据上述UMI-RNA-Seq的原理,我们可以发现UMI特异标签能够很好的消除文库构建过程中PCR扩增及测序仪引起的偏好性和重复性。但在测序过程中不止PCR扩增过程会产生干扰,逆转录突变、建库过程技术误差、测序数据量差异等都会导致转录组测序准确性下降,因此仅仅通过UMI标签不能完全消除这些差异,无法实现真正意义上的绝对定量测序。
云序生物UMI + spike-in矫正实现更精准定量测序!!
为了弥补UMI-RNA-Seq方法无法消除的逆转录突变、试验技术误差、测序结果数据量差异等缺点,云序生物联合UMI-RNA-Seq与spike-in矫正共同实现对转录组测序的精准绝对定量!
Spike-in是什么?
Spike in标准物,也称为外源性内标,是指在每个样品中加入等量的已知序列信息的非亲缘标准品基因组,与实验样品共同进行文库构建,这样在进行RNA-Seq测序时就可以通过不同样本之间内参(spike-in)量,非常准确地对不同样本之间的表达量进行矫正,很好的弥补UMI无法消除的偏差,从而实现测序高准确性定量。

a. 当基因组各处都发生相同程度的变化时,将总测序reads归一化为相同的数字会隐藏变化,而将spike-in的reads归一化为相同的数字会揭示reads密度的全局变化。
b. 当特定基因组区域发生信号增加时,标准化样本之间的总测序reads会导致来自基因组其他区域的reads数量人为减少,这就会错误地解释为在特定实验条件下减少。而通过使用spike-in的reads作为标准化,可以避免这种人为的变化。
技术优势

技术适用范围
-
适用于全转录组测序、mRNA 测序、LncRNA测序、circRNA 测序等多种RNA测序
-
适用于MeRIP-m6A--Seq 、MeRIP-m5C-Seq、acRIP-Seq 等多种 RNA 修饰测序
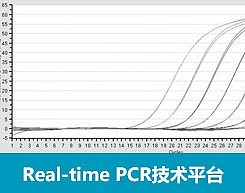
数据示例
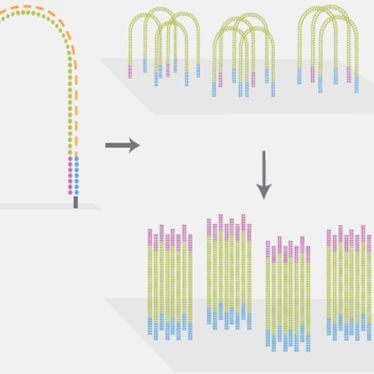
转录组测序结果示例
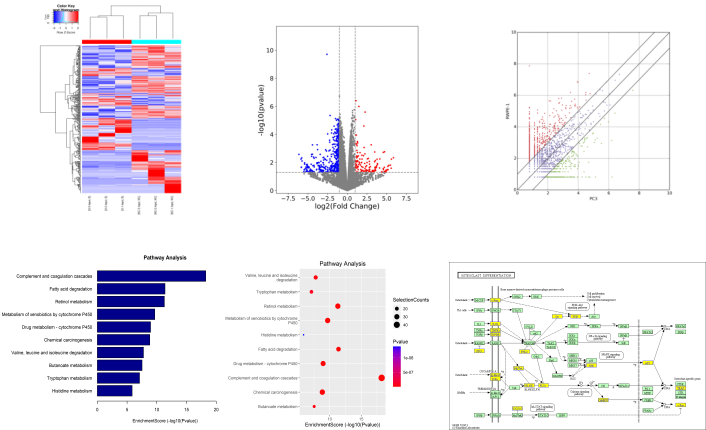
RNA修饰测序结果示例

为MeRIP实验的准确性及数据有效性提供了有力支持!
阳性(添加了相应修饰的spike in):检测到较高的信号值
阴性(添加了不带修饰的spike in):几乎检测不到信号值


上海云序生物科技有限公司
实名认证
钻石会员
入驻年限:10年