产品详情
文献和实验
相关推荐
保存条件 :-20
保质期 :2年
英文名 :pGAPZαB
库存 :100
供应商 :上海烜雅生物科技有限公司
CAS号 :/
规格 :干粉/液体
基本信息名称:pGAPZαB毕赤酵母质粒
别称: pGAPZαB
| 别称: | pGAPZaB |
|---|---|
| 启动子: | GAP |
| 复制子: | pUC |
| 终止子: | AOX1 TT |
| 质粒分类: | 酵母系列,毕赤酵母表达载体 |
| 质粒大小: | 3151bp |
| 质粒标签: | C-Myc, C-6×His |
| 原核抗性: | Zeo |
| 筛选标记: | Zeo |
| 克隆菌株: | DH5a |
| 培养条件: | 37度 |
| 表达宿主: | 酵母细胞 |
| 培养条件: | 28℃,YPD |
| 5'测序引物: | pGAP-F:GTCCCTATTTCAATCAATTGAA |
| 3'测序引物: | AOX3:GGCAAATGGCATTCTGACAT |
质粒属性
| 载体宿主: | 酵母菌 |
|---|---|
| 载体用途: | 蛋白表达 |
| 基因种属: | 空载体 |
| 基因类型: | ORF |
| 原核抗性: | Zeo |
| 筛选标记: | Zeo |
| 荧光蛋白: |
质粒简介
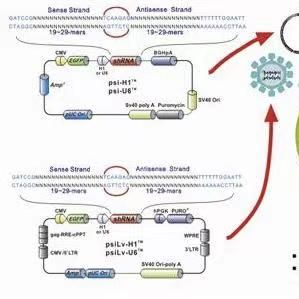
甘油醛-3-磷酸脱氢酶(GAPDH)的酶在许多生物细胞中包括毕赤酵母,稳定高水平表达。最近GAPDH蛋白编码基因启动子(GAP)被深入研究,并在毕赤酵母中能够高水表达重组蛋白,这取决于使用的碳源(Waterham等 人,,1997)。GAP启动子(PGAP)的重组蛋白表达水平略高于AOX1启动子。
pGAPZ A,B,和C载体(2.9 KB)和pGAPZαB和C(3.1 KB)载体使用GAP启动子在毕赤酵母中稳定表达重组蛋白。重组蛋白表达为为带有C-末端myc抗原表位和C端His标签的融合蛋白。此外,pGAPZα 表达的融合蛋白在N-端带有酿酒酵母(Saccharomyces cerevisiae)的α-因子分泌信号肽。这两个系列的载体都提供三个不同读码框的载体,以方便克隆的带有C-端标签和/或N-末端分泌信号肽的载体中。

质粒图谱

质粒序列
LOCUS Exported 3151 bp ds-DNA circular SYN 10-SEP-2016
DEFINITION synthetic circular DNA
ACCESSION .
VERSION .
KEYWORDS Untitled 13
SOURCE synthetic DNA construct
ORGANISM synthetic DNA construct
REFERENCE 1 (bases 1 to 3151)
AUTHORS .
TITLE Direct Submission
JOURNAL Exported Saturday, September 10, 2016 from SnapGene Viewer 3.1.4
http://www.miaolingbio.com
FEATURES Location/Qualifiers
source 1..3151
/organism="synthetic DNA construct"
/mol_type="other DNA"
promoter 7..483
/note="GAP promoter"
/note="promoter for Pichia pastoris
glyceraldehyde-3-phosphate dehydrogenase"
CDS 493..759
/codon_start=1
/gene="MF-alpha-1"
/product="N-terminal secretion signal from S. cerevisiae
alpha-factor"
/note="alpha-factor secretion signal"
/note="Cleavage by the Kex2 protease occurs after the
dibasic KR sequence. The EA dipeptides are then removed by
dipeptidyl aminopeptidase A."
/translation="MRFPSIFTAVLFAASSALAAPVNTTTEDETAQIPAEAVIGYSDLE
GDFDVAVLPFSNSTNNGLLFINTTIASIAAKEEGVSLEKREAEA"
CDS 831..860
/codon_start=1
/product="Myc (human c-Myc oncogene) epitope tag"
/note="Myc"
/translation="EQKLISEEDL"
CDS 876..893
/codon_start=1
/product="6xHis affinity tag"
/note="6xHis"
/translation="HHHHHH"
terminator 972..1218
/gene="Pichia pastoris AOX1"
/note="AOX1 terminator"
/note="transcription terminator for AOX1"
promoter 1233..1644
/gene="S. cerevisiae TEF1"
/note="TEF1 promoter"
/note="promoter for EF-1-alpha"
promoter 1651..1698
/note="EM7 promoter"
/note="synthetic bacterial promoter "
CDS 1717..2091
/codon_start=1
/gene="Sh ble from Streptoalloteichus hindustanus"
/product="antibiotic-binding protein"
/note="BleoR"
/note="confers resistance to bleomycin, phleomycin, and
Zeocin(TM)"
/translation="MAKLTSAVPVLTARDVAGAVEFWTDRLGFSRDFVEDDFAGVVRDD
VTLFISAVQDQVVPDNTLAWVWVRGLDELYAEWSEVVSTNFRDASGPAMTEIGEQPWGR
EFALRDPAGNCVHFVAEEQD"
terminator 2157..2404
/gene="S. cerevisiae CYC1"
/note="CYC1 terminator"
/note="transcription terminator for CYC1"
rep_origin complement(2479..3067)
/direction=LEFT
/note="ori"
/note="high-copy-number ColE1/pMB1/pBR322/pUC origin of
replication"
ORIGIN
1 agatcttttt tgtagaaatg tcttggtgtc ctcgtccaat caggtagcca tctctgaaat
61 atctggctcc gttgcaactc cgaacgacct gctggcaacg taaaattctc cggggtaaaa
121 cttaaatgtg gagtaatgga accagaaacg tctcttccct tctctctcct tccaccgccc
181 gttaccgtcc ctaggaaatt ttactctgct ggagagcttc ttctacggcc cccttgcagc
241 aatgctcttc ccagcattac gttgcgggta aaacggaggt cgtgtacccg acctagcagc
301 ccagggatgg aaaagtcccg gccgtcgctg gcaataatag cgggcggacg catgtcatga
361 gattattgga aaccaccaga atcgaatata aaaggcgaac acctttccca attttggttt
421 ctcctgaccc aaagacttta aatttaattt atttgtccct atttcaatca attgaacaac
481 tatttcgaaa cgatgagatt tccttcaatt tttactgctg ttttattcgc agcatcctcc
541 gcattagctg ctccagtcaa cactacaaca gaagatgaaa cggcacaaat tccggctgaa
601 gctgtcatcg gttactcaga tttagaaggg gatttcgatg ttgctgtttt gccattttcc
661 aacagcacaa ataacgggtt attgtttata aatactacta ttgccagcat tgctgctaaa
721 gaagaagggg tatctctcga gaaaagagag gctgaagctg caggaattca cgtggcccag
781 ccggccgtct cggatcggta cctcgagccg cggcggccgc cagctttcta gaacaaaaac
841 tcatctcaga agaggatctg aatagcgccg tcgaccatca tcatcatcat cattgagttt
901 tagccttaga catgactgtt cctcagttca agttgggcac ttacgagaag accggtcttg
961 ctagattcta atcaagagga tgtcagaatg ccatttgcct gagagatgca ggcttcattt
1021 ttgatacttt tttatttgta acctatatag tataggattt tttttgtcat tttgtttctt
1081 ctcgtacgag cttgctcctg atcagcctat ctcgcagctg atgaatatct tgtggtaggg
1141 gtttgggaaa atcattcgag tttgatgttt ttcttggtat ttcccactcc tcttcagagt
1201 acagaagatt aagtgagacc ttcgtttgtg cggatccccc acacaccata gcttcaaaat
1261 gtttctactc cttttttact cttccagatt ttctcggact ccgcgcatcg ccgtaccact
1321 tcaaaacacc caagcacagc atactaaatt ttccctcttt cttcctctag ggtgtcgtta
1381 attacccgta ctaaaggttt ggaaaagaaa aaagagaccg cctcgtttct ttttcttcgt
1441 cgaaaaaggc aataaaaatt tttatcacgt ttctttttct tgaaattttt ttttttagtt
1501 tttttctctt tcagtgacct ccattgatat ttaagttaat aaacggtctt caatttctca
1561 agtttcagtt tcatttttct tgttctatta caactttttt tacttcttgt tcattagaaa
1621 gaaagcatag caatctaatc taagggcggt gttgacaatt aatcatcggc atagtatatc
1681 ggcatagtat aatacgacaa ggtgaggaac taaaccatgg ccaagttgac cagtgccgtt
1741 ccggtgctca ccgcgcgcga cgtcgccgga gcggtcgagt tctggaccga ccggctcggg
1801 ttctcccggg acttcgtgga ggacgacttc gccggtgtgg tccgggacga cgtgaccctg
1861 ttcatcagcg cggtccagga ccaggtggtg ccggacaaca ccctggcctg ggtgtgggtg
1921 cgcggcctgg acgagctgta cgccgagtgg tcggaggtcg tgtccacgaa cttccgggac
1981 gcctccgggc cggccatgac cgagatcggc gagcagccgt gggggcggga gttcgccctg
2041 cgcgacccgg ccggcaactg cgtgcacttc gtggccgagg agcaggactg acacgtccga
2101 cggcggccca cgggtcccag gcctcggaga tccgtccccc ttttcctttg tcgatatcat
2161 gtaattagtt atgtcacgct tacattcacg ccctcccccc acatccgctc taaccgaaaa
2221 ggaaggagtt agacaacctg aagtctaggt ccctatttat ttttttatag ttatgttagt
2281 attaagaacg ttatttatat ttcaaatttt tctttttttt ctgtacagac gcgtgtacgc
2341 atgtaacatt atactgaaaa ccttgcttga gaaggttttg ggacgctcga aggctttaat
2401 ttgcaagctg gagaccaaca tgtgagcaaa aggccagcaa aaggccagga accgtaaaaa
2461 ggccgcgttg ctggcgtttt tccataggct ccgcccccct gacgagcatc acaaaaatcg
2521 acgctcaagt cagaggtggc gaaacccgac aggactataa agataccagg cgtttccccc
2581 tggaagctcc ctcgtgcgct ctcctgttcc gaccctgccg cttaccggat acctgtccgc
2641 ctttctccct tcgggaagcg tggcgctttc tcaatgctca cgctgtaggt atctcagttc
2701 ggtgtaggtc gttcgctcca agctgggctg tgtgcacgaa ccccccgttc agcccgaccg
2761 ctgcgcctta tccggtaact atcgtcttga gtccaacccg gtaagacacg acttatcgcc
2821 actggcagca gccactggta acaggattag cagagcgagg tatgtaggcg gtgctacaga
2881 gttcttgaag tggtggccta actacggcta cactagaagg acagtatttg gtatctgcgc
2941 tctgctgaag ccagttacct tcggaaaaag agttggtagc tcttgatccg gcaaacaaac
3001 caccgctggt agcggtggtt tttttgtttg caagcagcag attacgcgca gaaaaaaagg
3061 atctcaagaa gatcctttga tcttttctac ggggtctgac gctcagtgga acgaaaactc
3121 acgttaaggg attttggtca tgcatgagat c
//
质粒菌株产品操作说明书
一、扩增流程
收到产品后,请先根据产品管壁标签来判断产品形式,并在扩增前准确查找该质粒菌株的抗性、感受态和培养温度。
1、质粒干粉(常温运输,存于-20度,90天保质期,请务必转化挑单克隆培养,不要直接使用和测序)
①收到质粒干粉后请先5000rpm离心1min,再加入20μl ddH2O去离子水溶解质粒;
②取1支100μl 感受态于冰上解冻10min,加入2μl质粒,再冰浴30min后,42℃热激60s,不要搅动,再冰浴2min;(从第二步开始均要在超净工作台中无菌操作)
③加入900μl无抗的LB液体培养基,180rpm震荡37℃培养45min;
④6000rpm离心5min,仅留100μl上清液重悬细菌沉淀,并涂布至目标质粒抗性的LB平板上;(可使用本平台的平板涂布专用玻璃珠进行涂布,可以比传统涂布方法获得更多转化子)
⑤将平板正向培养1h,再倒置37℃培养14h。如果要求是30度则培养20h;
(菌落过多则将质粒稀释后再转化。没有菌落则加入10μl质粒转化。另不要直接转表达感受态,要先转克隆感受态,重提质粒后再导入表达感受态)
⑥挑取单菌落至LB液体培养基中,加入对应抗生素,220rpm震荡培养14h,根据实验需要和质粒提取试剂盒说明书提取质粒。
2、甘油菌种(冰袋运输,存于-80℃,保质期90天,请务必划线挑单克隆培养)
四区划线后挑单菌落培养,酵母菌需要先液体复苏再四区划线,再挑单菌落液体培养。
3、穿刺菌种(冰袋运输,存于4℃,保质期7天)
穿刺接种,液体培养后四区划线,再挑单菌落液体培养。
4、菌落平板(冰袋运输,存于4℃,保质期7天)
直接挑取单菌落至液体培养基中。
5、液体质粒(冰袋运输,存于-20℃,保质期90天)
单独提取的液体质粒收到后可直接使用。
6、滤纸质粒(常温运输,存于-20度,90天保质期,请务必转化挑单克隆培养,不要直接使用和测序)
收到货后将滤纸画圈部分剪下放入EP管中,加100ul无菌水将滤纸浸湿并浸泡5min,吸取5ul质粒转化,离心全涂。
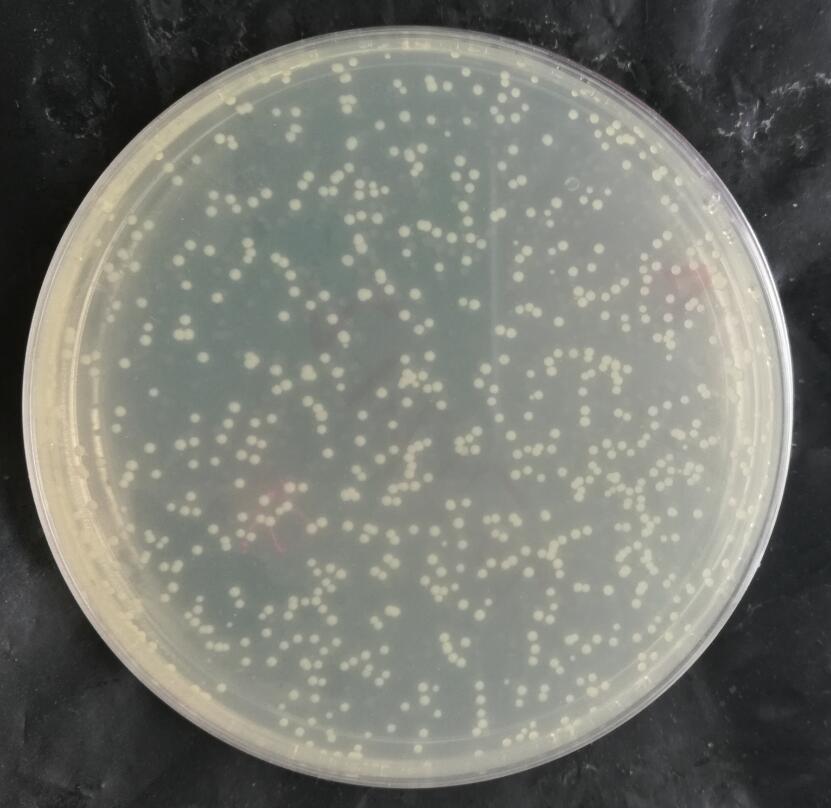
二、转化图片

| P4526/pLenti-CMV-MCS-GFP-SV-puro慢病毒表达质粒 |
| P4868/pWPI病毒表达质粒 |
| P0820/pWPXL慢病毒表达质粒 |
| P0822/pWPXLd慢病毒表达质粒 |
| P0882/pLEGFP-N1慢病毒表达质粒 |
| P0921/pL-SIN-PGK-EGFP慢病毒表达质粒 |
| P0922/pLVPT-tTR-KRAB慢病毒表达质粒 |
| P0923/pLVPT-rtTR-KRAB-2SM2慢病毒表达质粒 |
| P0924/pHR_Gal4UAS_IRES_mC_pGK_tBFP 慢病毒表达质粒 |
| P0981/pGZ慢病毒表达质粒 |
| P0982/pLEX-MCS慢病毒表达质粒 |
| P0983/pPACKH1-REV慢病毒表达质粒 |

上海烜雅生物科技有限公司
实名认证
钻石会员
入驻年限:7年