非编码 RNA 可火了,miRNA 大火烧了十几年,现在慢慢地把 lncRNA 的领域也给点着了。现在可能连实验室的插枪头阿姨都知道绝大多数的 RNA 分子并不翻译,这非编码 RNA(noncoding RNA),包括 miRNA、lncRNA 等。
目前主要研究方向集中在非编码 RNA 及其相互结合的 DNA、RNA、蛋白质等科学问题,而科学问题的解答依赖于实验技术的创新使用和革命。
ncRNA 可以海陆空全面调控细胞功能,厉害了,其实主要指 lncRNA,而 miRNA、siRNA 主要作用于 mRNA 层面,本文按非编码 RNA 的三个调控维度,就当前主要的实验技术简单介绍一下。
一、DNA 维度
作用的 ncRNA 类型:lncRNA 等
研究方法介绍有:
和 DNA 序列结合的调控目的无非是①阻止下游基因的转录②把转录因子、甲基化酶等招惹过来修饰 DNA 序列。他们的共同特点是和解链的 DNA 的某一条链结合,那么研究方法包括
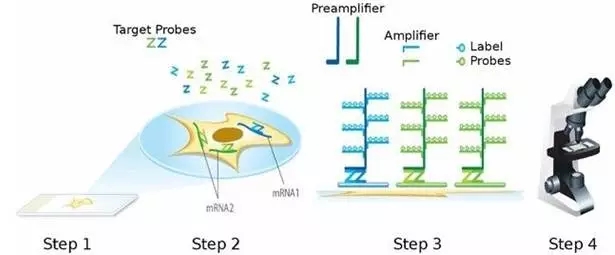
1. RNA 原位杂交 (RNAin situ hybridization)
检测该 ncRNA 是否位于核内,如果不在核内,那就不用考虑和 DNA 作用了。
RNA 原位杂交是指将特定标记的已知顺序核酸为探针与细胞或组织切片中核酸进行杂交,从而对特定核酸顺序进行精确定量定位的过程。原位杂交可以在细胞标本或组织标本上进行。根据 RNA 序列设计 cDNA、cRNA 探针,对目标进行定位。
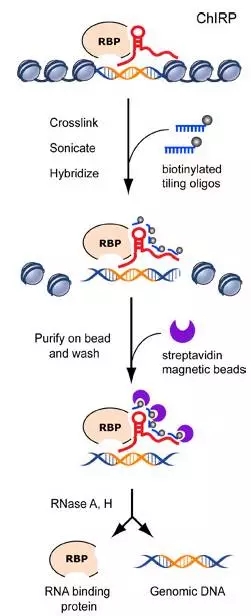
2. ChIRP(chromatin isolation by RNA purifications)
是一项通过纯化 RNA 分子从而获得其结合的染色质片段 ( 包括 RNA 结合蛋白与基因组 DNA) 的实验方法。
大概流程:首先细胞交联,使 lncRNA、染色质及被固定连接;随后通过超声将染色质打断,并使用带有生物素标记的 tiling oligos 与长链非编码 RNA 分子杂交;之后利用亲和素磁珠纯化长链非编码 RNA 及其结合的染色质片段;后续分析和测序
3. ncRNA-DNA 三联体的体外同位素检测
为了进一步检验这种调控机制和结合方式,可以在体外分别合成此 ncRNA 和 DNA 片段,并用同位素(P32)标记,在体外做结合实验,如果不结合,那么就会呈现两条已知的条带,如果能够结合,那么就会有一条 RNA-DNA 分子量新条带出现。
二、RNA 维度
作用的 ncRNA 类型:mRNA-miRNA-lncRNA+circRNA 等
研究方法
既然是 RNA-RNA 相互作用,除了胞内 FISH 定位技术以外,还有其他技术要使用,如下
1. CLASH (crosslinking-ligation and sequencing ofhybrids)
对 RNA 分子与 RNA 分子相互作用进行研究的实验系统,主要被用于鉴定 miRNA 与其靶 mRNA 的相互作用。大概流程为:首先对细胞进行交联处理,将 miRNA 及其靶 mRNA 与 RISC 复合物固定连接;随后通过 Co-IP 富集纯化 RISC 复合物;之后再通过分子内连接将 miRNA 的 3'-OH 末端与靶 mRNA 的 5'-PO4 末端连接,形成一条长的单链 RNA,然后测序。
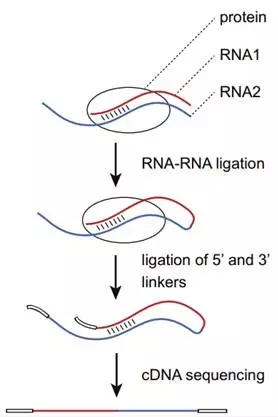
2. CHART(CaptureHybridization Analysis of RNA Targets)
也可以叫做 RNA precipitation,根据 ncRNA 序列设计探针,二次探针交联在固相载体上,若将核内 RNA-DNA 状态固定处理,染色质超声打碎成小片段,RNA-DNA 的结构就可以被探针抓取下来,利用 DNA-Seq 对抓取的 DNA 进行测序,获得 DNA 结合序列的信息(不过这个可以被生信分析工具所预测一下)。
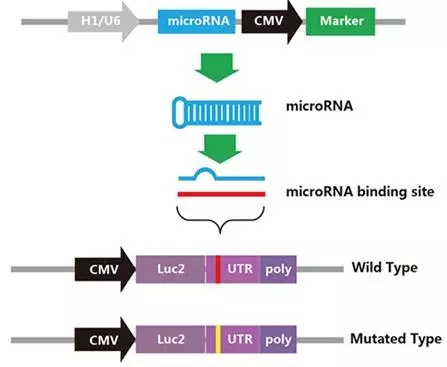
3. 荧光素酶报告系统
在荧光素酶报告基因载体的报告基因编码区的下游插入 ncRNA 结合序列或者 mRNA 的 3』UTR 区,然后将构建好的载体转染细胞并改变细胞中相应 miRNA 的表达水平,通过检测荧光素酶的表达情况以分析 ncRNA 或者 mRNA 的 3』UTR 区中是否含有 miRNA 的靶位点。同理任意两种 RNA-RNA 互作都可以利用这个原理。
三、蛋白质维度
作用的 ncRNA 类型:lncRNA、mRNA 较多
研究方法:
这种互作模式是 RNA-Protein,那么 ncRNA 可以和调控 DNA 的蛋白结合,也可以和细胞质内的蛋白质结合,发挥不同效应。
1. CLIP-seq:鉴定与特定 RNA 结合蛋白相互作用的 RNA 分子
CLIP-seq 也被称为 HITS-CLIP,是将紫外交联免疫共沉淀技术 (CLIP) 与高通量测序技术相结合的技术。
大概流程为:首先通过紫外照射将细胞内的 RNA 分子与 RNA 结合蛋白进行耦联;细胞裂解,免疫共沉淀,核酸酶 (RNase) 将共沉淀的 RNA 切割,只保留蛋白质内部的 RNA 片段;通过 5' 末端放射性标记 RNA 分子及 SDS-PAGE 电泳,选择性地回收目的蛋白结合的 RNA 片段;最后高通量测序。
其可以衍生了许多变异的 CLIP-seq 方法,例如增强了交联强度的 PAR-CLIP 和能够精确鉴定 RNA 分子中蛋白质结合位点的 iCLIP 等。

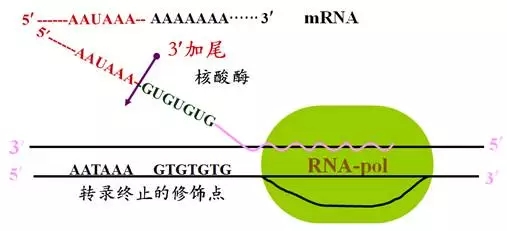
2. mRNA-蛋白研究方法:利用 Poly(A) 尾
首先紫外交联 (UV-CL),将 mRNA 与蛋白质的相互作用固定连接;随后使用耦联磁珠的 Oligo(dT) 富集纯化 mRNA 蛋白复合物,经过洗涤及降解 RNA 等步骤,最后将获得的蛋白质进行质谱分析。
3. EMSA
凝胶迁移或电泳迁移率实验(EMSA-electrophoretic mobility shift assay)用于研究 RNA 结合蛋白和特定的 RNA 序列的相互作用。
通常将纯化的蛋白和细胞粗提液和 32P 同位素标记的 RNA 探针一同保温,在非变性的聚丙烯凝胶电泳上,分离复合物和非结合的探针。RNA-复合物比非结合的探针移动得慢。
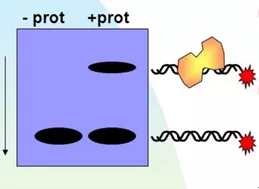
4.RIP(RNA-immuno precipitation)
RNA 免疫沉淀可以用来研究组织或活细胞中蛋白质和 RNA 之间的相互作用。该方法的原理是采用针对目标蛋白的抗体, 把相应的 RNA-蛋白复合物沉淀下来, 经过分离纯化, 并对复合物上的 RNA 进行分析鉴定。
RIP 技术分别与 Chip 和 RNA-Seq 相结合而衍生的 RIP-Chip 和 RIP-Seq 技术, 也广泛应用于 lncRNA 与蛋白质的相互作用中。



你可能喜欢